Dinámica de proteínas
Función dinámica
Las proteínas globulares tienen en común el unirse a otra molécula más pequeña llamada ligando para ser funcionales. Hay un sitio de unión perfectamente definido donde se une el ligando de manera específica y selectiva. Es una parte pequeña de la proteína pero esencial. Este sitio tendrá grupos funcionales que establezcan uniones de tipo débil con el ligando y deberá ser accesible desde fuera. Normalmente el sitio de unión y el ligando son complementarios, sólo cabe ése y no otro. Se forma un complejo proteína - ligando que es reversible excepto en el caso de antígenos y anticuerpos. Si la proteína es un enzima catalizará la reacción en que el ligando es el sustrato, la proteína se une al ligando que se transforma en producto y luego se disocia y la enzima queda libre. La función se ejerce sobre el ligando.
Consideraciones generales sobre la interacción proteína - ligando
Función de saturación.
1 sitio de unión por proteína
| P + L ⇌ PL: Kₘ = | [P]·[L] |
| [PL] |
K es en el sentido de disociación del ligando. La actividad biológica depende de cuanto ligando hay unido, por lo tanto, de la concentración de ligando.
La función de saturación υ son los moles de ligando unidos partido por los moles de proteína totales:
| v = | [PL] | = | [L] |
| [P] + [PL] | K + [L] |
El valor máximo de la función de saturación es 1, como depende de la concentración de ligando, cuanto más haya mayor será υ. Es la ecuación de una hipérbola con una asíntota en 1:
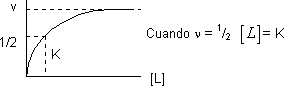
Transformando en una recta:
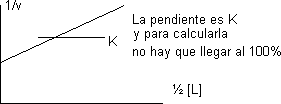
Más de un sitio de unión por proteína
Se ocupan con el mismo ligando. Si tiene 2 sitios de unión se ocupa primero uno y luego el otro. Si todos los sitios son iguales (K iguales) hay dos posibilidades:
Sitios independientes:
Es el caso más sencillo. Si hay varios sitios iguales es porque la proteína tiene varias cadenas iguales con un sitio cada una. Como son iguales K₁ = K₂ y no se puede saber cual se ocupa primero, son independientes porque no hay conexión entre ellos. La ocupación de un sitio tiene la misma constante aunque ya haya otro ocupado.
Si la proteína tiene dos sitios de unión:
| v = | [PL] + [LP] + 2·[PL₂] |
| [P] + [PL] + [LP] + [PL₂] |
| v = | ∑[L] | La asíntota estará en 2 |
| K' + [L] |
4 Para compara proteína se usa la fracción de saturación: Y = υ /n
Ahora la variación es entre 0 y 1. Sigue siendo una hipérbola.
El porcentaje de saturación es Y×100. Fracción y porcentaje de saturación van unidos a la funcionalidad de la proteína.
Sitios que interaccionan:
Se produce un efecto llamado cooperatividad en el que cuando se ocupa un sitio activo la unión de un segundo ligando en un sitio igual se ve afectada, siendo favorecida (cooperatividad positiva) o perjudicada (cooperatividad negativa). Para que haya cooperatividad se deben dar una serie de requisitos:
Deben haber varios sitios iguales, por lo tanto, varias cadenas iguales, la proteína será oligomérica. Todas las oligoméricas presentan cooperatividad, la mayor parte de las veces positiva. Ahora υ no es una hipérbola sino una curva sigmoidea:
| v = | n·[L]ᶜ |
| K + [L]ᶜ |
En el caso de la unión del O₂ por la hemoglobina hay cuatro sitios. Las proteínas tienen poca afinidad por el ligando al principio pero como presenta cooperatividad positiva cada vez se unen más rápido y se obtiene un salto en la curva.
Explicación de la curva sigmoidea por Hill:
Si la cooperatividad fuera infinita en cuanto se uniera un ligando inmediatamente se unen los demás, la proteína tendría una afinidad muy alta por el ligando.
| v = | n·[L]ᶜ |
| K + [L]ᶜ |
c es el coeficiente de Hill que cuantifica la cooperatividad. Si c = 1 no hay cooperatividad. La cooperatividad será positiva si c varía entre 1 y n y negativa si varía entre 0 y 1. Las proteínas cooperativas al representar υ frente a [L] sale una curva sigmoidea con asíntota en n, al representar Y frente a [L] la asíntota estará en 1, y si son porcentajes la asíntota es 100.
Las proteínas cooperativas son más sensibles a los cambios en la concentración de ligando porque para conseguir una variación en la saturación hay que variar menos la concentración de sustrato porque la entrada de uno favorece las de los demás. Esto es importante para la célula porque ahorra espacio.
Efecto homotrópico: efecto de la entrada de un ligando sobre la entrada de otro igual. La mayor parte de las proteínas cuya función hay que controlar son cooperativas en la unión del ligando son casi siempre oligoméricas.
Efecto heterotrópico: efecto de un ligando sobre otro distinto. Tienen otros sitios específicos distintos para unir ligandos distintos llamados moduladores o efectores. La acción no se lleva a cabo sobre él sino que puede afectar a la actividad de la proteína de 2 maneras distintas:
- Si el ligando se une mejor: el modulador es un activador que se une en un sitio distinto del activo y favorece la unión del ligando
- Si el ligando se une peor: el modulador es un inhibidor que perjudica la unión del ligando
Este efecto se llama alosterismo. Las proteínas que es necesario regular presentan cooperatividad en la unión del ligando y alosterismo porque son dos procesos muy ventajosos. Para que haya alosterismo la proteína no debe ser oligomérica necesariamente Al actuar un activador la curva sigmoidea se desplaza a la izquierda y el límite es la hipérbola, la cooperatividad baja porque mejora la afinidad por el ligando (el efecto del sustrato es menor) y en la hipérbola es nula. Al actuar un inhibidor la curva se desplaza a la derecha.
Explicación estructural de la cooperatividad y el alosterismo
Cuando el ligando se une se produce un cambio en la conformación.
Cooperatividad: al unirse el ligando la proteína se acomoda a él produciendo un pequeño cambio en la conformación. Si la proteína presenta estructura cuaternaria las interacciones entre las subunidades cambian, con lo que varía la conformación. Si es mejor que la anterior la cooperatividad es positiva y si no negativa. Si se pierde la estructura cuaternaria se desunen las subunidades y al no comunicarse se pierde la cooperatividad.
Alosterismo: igual para el activador, en el caso del inhibidor se dificulta la entrada del ligando.
Modelos moleculares
Modelo concertado (Jacques-Lucien Monod, Wyman, Changeux)
Propone que la proteína puede existir en dos conformaciones, la forma T (tensa) con poca afinidad por el ligando y la forma R (relajada) con mucha afinidad. Las dos formas existen en ausencia de ligando y la proteína es simétrica, cada subunidad no puede tener conformación distinta a la otra. Todo lo que favorezca la forma R tendrá un efecto positivo sobre la unión del ligando. Al añadir más ligando se va estabilizando la forma R y cada vez hay más. En la cooperación positiva el ligando se une a la forma R y la estabiliza para que no vuelva a la forma T (como un activador) y en la negativa se estabiliza la forma T.
Constante alostérica: relación entre la forma de baja afinidad y la de alta. Cuanta más cooperatividad hay más vale L.
L = [T]/[R]
Modelo secuencial (Koshland, Nametry, Filmer)
La proteína puede existir en dos conformaciones, una de alta afinidad por el ligando y otra de baja. El cambio en la conformación está inducido por la entrada del sustrato. Al unirse el ligando cambia la conformación de la otra subunidad, afectando a las interacciones entre las subunidades y permite la unión de otro ligando. Si K₂ > K₁ el efecto es positivo y si es al revés es negativo.
Editor: Ricardo Santiago Netto (Administrador de Fisicanet).